L’équipe
Fabien LE GRAND
DR2 CNRS, HDR
0000-0002-7843-3899
Pauline GARCIA
PhD Student
0000-0002-4673-8583
Sidy FALL
IE, Bioinformaticien
Fany AGOSTINI
Assistante Ingénieur
0000-0001-5569-4917
Jade RAVENT
Technicienne supérieure
0000-0002-5023-4230
Julie SITOLLE
PhD Student
Légende :
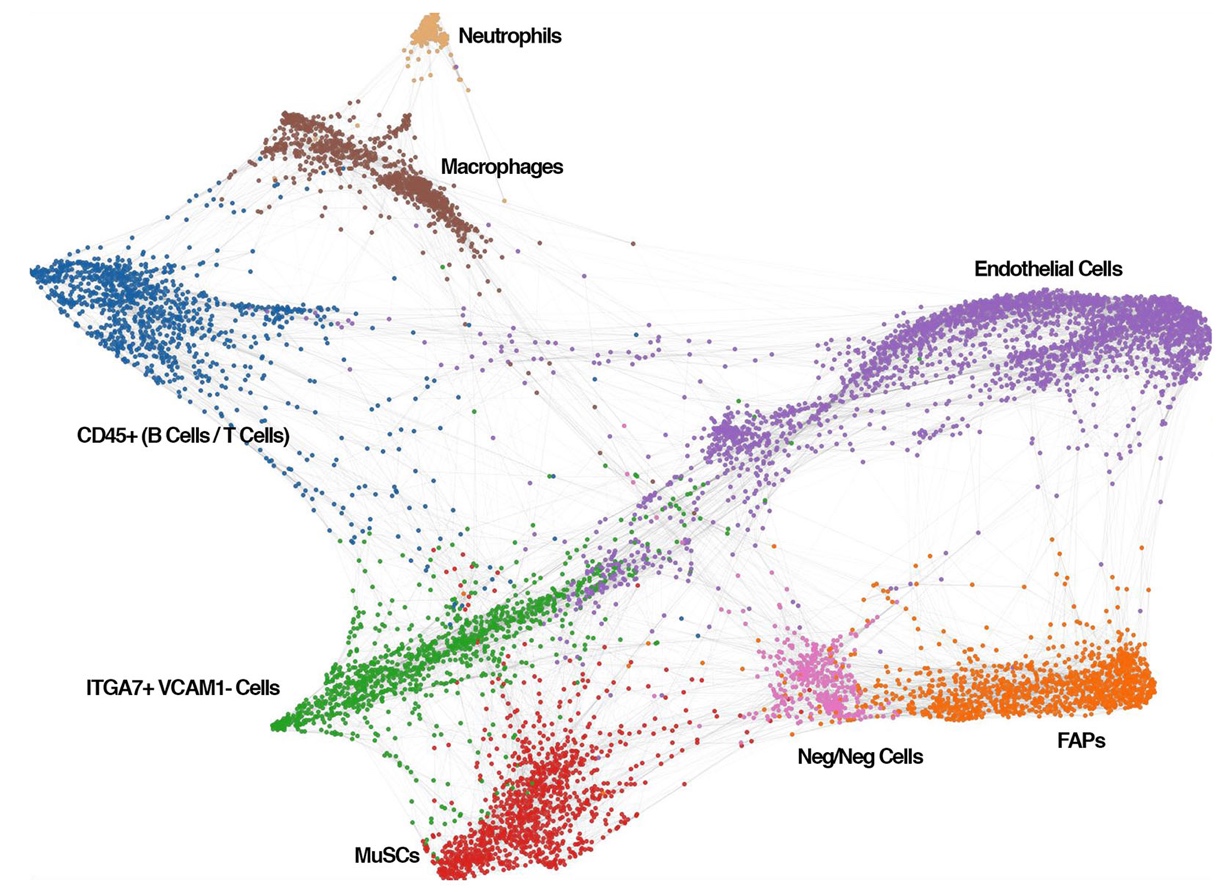
Afin de caractériser la diversité cellulaire du muscle squelettique, nous avons réalisé une expérience de cytométrie de masse (CyTOF) sur des suspensions cellulaires mononucléaires provenant de muscles postérieures de la souris adulte. Nous montrons ici l’analyse des données de CyTOF avec une visualisation orientée en cellule unique. Chaque cluster a été annoté selon l’expression de gènes marqueurs de différents types cellulaires majoritaires. Les clusters partageant les mêmes gènes marqueurs canoniques sont représentés avec la même couleur.
Projets
La régénération des muscles squelettiques repose sur les interactions orchestrées entre les cellules souches musculaires et les autres cellules qui résident ou envahissent le tissu blessé. Ces interactions, médiées par des voies de signalisation, contrôlent les fonctions et les transformations phénotypiques des cellules souches en réponse aux signaux environnementaux.
Des perturbations dans l’architecture de cet hyper-réseau mènent généralement à des défauts de régénération. De plus, les composantes nécessaires à la construction de la niche « régénérative » des cellules souches sont mal connus. L’équipe SPaSM réunit des expertises en biologie fondamentale et en recherche translationnelle pour comprendre la régénération musculaires par une approche multi-échelle. Nous nous concentrons sur les voies de signalisation déployées au cours de la régénération du muscle squelettique dans le but de développer de nouvelles stratégies thérapeutiques pour les dystrophies musculaires.
Notre recherche s’articule autour de trois objectifs :
i/ En utilisant des technologies cellules uniques dit « single cell », nous cartographions la composition cellulaire des muscles striées (analyses omiques : séquençage de l’ARN, disponibilité de la chromatine, analyses des interactions protéines-ADN, cytométrie de masse) au cours du développement, de la régénération, du vieillissement et dans les modèles murins de dystrophies musculaires, notamment la Dystrophie de Duchenne.
ii/ Nous étudions la dynamique du cytosquelette suite à la modulations de voies de signalisation extracellulaire pour comprendre les signaux qui régulent la fusion des myoblastes squelettiques.
iii/ Nous avons observé les signaux Wnt/ß-caténine et Tgfß étaient sur-activés dans le tissu musculaire au cours du vieillissement et dans les dystrophies musculaires. Nous déchiffrons à présent les conséquences fonctionnelles de ces perturbations.
FINANCEMENTS
- ANR Myocity (ANR-19-RAR4-0015)
- ANR Myofuge
- ANR Myofibrosis
- AFM Strat2

![]()



Sélection de publications
- Le Grand F, Jones AE, Seale V, Scimè A, Rudnicki MA. Wnt7a activates the planar cell polarity pathway to drive the symmetric expansion of satellite stem cells. Cell Stem Cell.2009; 4(6) : 535-47.
- Le Grand F, Grifone R, Mourikis P, Houbron C, Gigaud C, Pujol J, Maillet M, Pagès G, Rudnicki M, Tajbakhsh S, Maire P. Six1 regulates stem cell repair potential and self-renewal during skeletal muscle regeneration. The Journal of Cell Biology. 2012;198(5):815-32.
- Parisi A, Lacour F, Giordani L, Colnot S, Maire P, Le Grand F. APC is required for muscle stem cell proliferation and skeletal muscle tissue repair. The Journal of Cell Biology.2015 Aug 31;210(5):717-26.
- Rudolf A, Schirwis E, Giordani L, Parisi A, Lepper C, Taketo MM, Le Grand F.β-Catenin Activation in Muscle Progenitor Cells Regulates Tissue Repair. Cell Reports. 2016 May 10;15(6):1277-90.
- Beyer S, Pontis J, Schirwis E, Battisti V, Rudolf A, Le Grand F*, Ait-Si-Ali S*. Canonical Wnt signaling regulates nuclear export of Setdb1 suring muscle terminal differentiation. Cell Discovery. 2016 Oct 17;2:16037. * equal contributions.
- Lacour F, Vezin E, Bentzinger CF, Sincennes MC, Giordani L, Ferry A, Mitchell R, Patel K, Rudnicki MA, Chaboissier MC, Chassot AA, Le Grand F. R-spondin1 Controls Muscle Cell Fusion through Dual Regulation of Antagonistic Wnt Signaling Pathways. Cell Reports.2017 Mar 7;18(10):2320-2330.
- Giordani, L., He, G.J., Negroni, E., Sakai, H., Law, J.Y., Siu, M.M., Wan, R., Tajbakhsh, S., Cheung, T.H., and Le Grand, F. High-dimensional single-cell cartography reveals novel skeletal muscle resident cell populations. Molecular Cell.2019 10.1016/j.molcel.2019.02.02
- Girardi F, Taleb A, Ebrahimi M, Datye A, G. Gamage D, Peccate C, Giordani L, P. Millay D, M. Gilbert P, Cadot B & Le Grand, F. TGFβ signaling curbs cell fusion and muscle regeneration. Nat Commun 12, 750 (2021). https://doi.org/10.1038/s41467-020-20289-8
Address
Institut NeuroMyoGène UCBL – CNRS UMR 5261 – INSERM U1315
Faculté de Médecine et de Pharmacie –
3ème étage – Aile B
8 avenue Rockefeller
69008 Lyon France