Assemblage des cils et développement
Chef d'équipe: Bénédicte DURAND
Cilia | Centriole | Cytoskeleton | Cell architecture | Ciliopathies | Drosophila | Microtubules Muscle | Nesprin
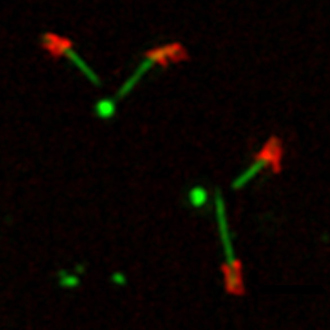
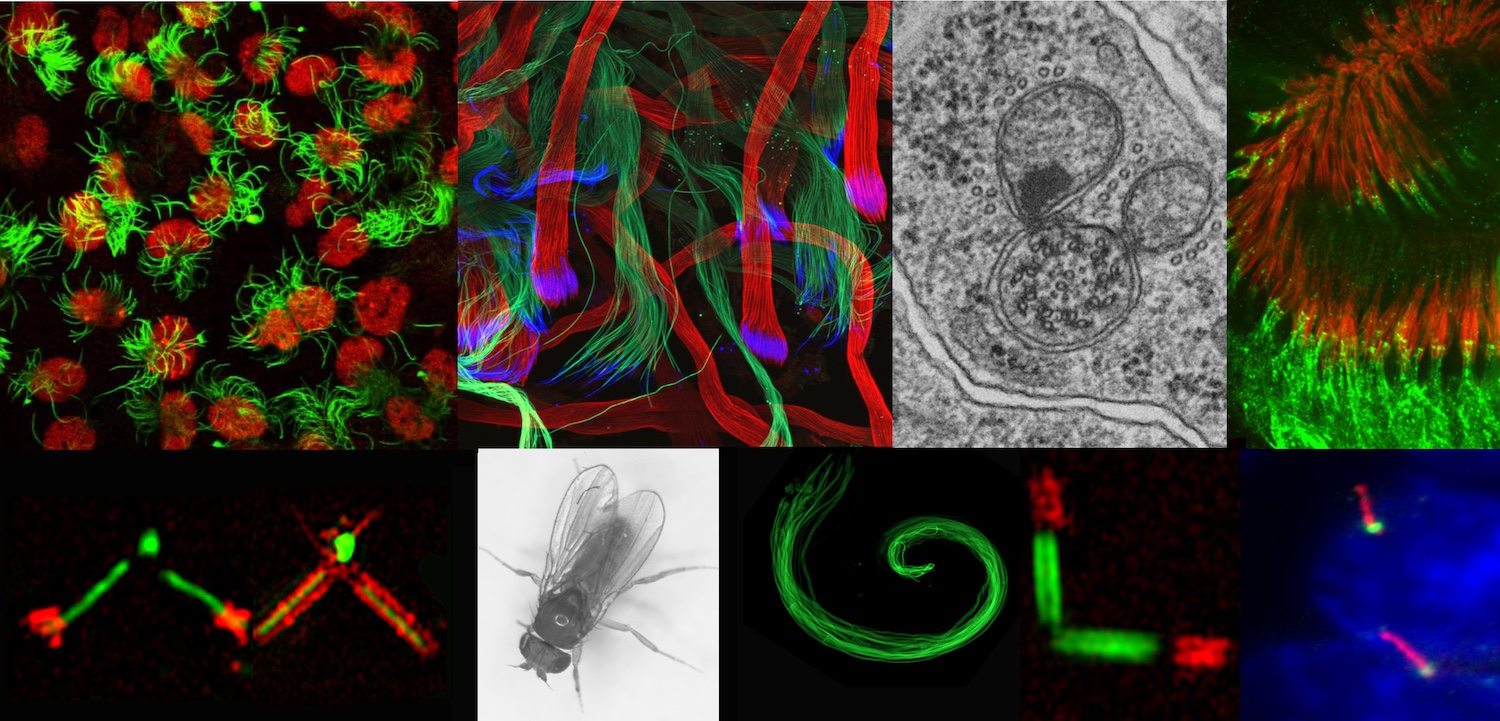
Une des étapes clés de l’assemblage des cils est la conversion du centriole en corps basal, processus strictement corrélé à la progression du cycle cellulaire. Un des défis actuels de la compréhension de l’assemblage des cils et des flagelles est notre capacité de résoudre l’organisation moléculaire de cette structure et d’identifier le rôle de chacun des constituants de cet assemblage hautement organisé. Pour cela des approches de microscopie haute résolution sont développées dans l’équipe : airyscan, 3D-SIM, microscopie électronique et microscopie d’expansion couplée à de la microscopie STED, appliquées à des tissus ciliés de drosophile ou des cellules de mammifère en culture afin de comprendre l’organisation moléculaire de la base des cils et la hiérarchie fonctionnelle d’assemblage de ses différents éléments constitutifs. Nous couplons ces capacités d’observations avec des outils de génétique fonctionnelle (dont édition du génome par CRISPR/Cas9). De plus nous développons des approches de biochimie (étiquetage de proximité) afin de comprendre la composition moléculaire des différents échafaudages ciliaires ou centriolaires. Grâce à ces approches combinées, nous avons déjà identifié plusieurs protéines qui sont impliquées chez l’Homme dans des pathologies ciliaires dont certaines sont également associées à des dégénérescences neuronales (sclérose latérale amyotrophique) et des pathologies du muscle cardiaque (cardiomyopathie). Nous cherchons à comprendre comment ces protéines contribuent à la biogenèse des cils et des centrioles et peuvent être à l’origine de ces différentes pathologies.
Pour le grand Public
Les cils et les flagelles sont de petites protrusions à la surface des cellules qui jouent des rôles variés : ils peuvent être mobiles et propulser des cellules (spermatozoïdes) ou des fluides (mucus respiratoire), ou immobiles et endosser alors le rôle d’antenne captant les signaux ambiants permettant à la cellule de s’adapter à son environnement. Des anomalies de formation ou de la fonction des cils sont responsables de nombreuses maladies héréditaires rares et souvent graves, appelées ciliopathies. Par ailleurs, les cils et leurs fondations, les centrioles, sont également des régulateurs importants des processus cancéreux en raison de leur rôle dans le contrôle de la division cellulaire et dans la communication entre cellules.
Notre équipe cherche à comprendre comment les cils sont assemblés à partir des centrioles et comment des variations autour d’un assemblage très conservé sont à l’origine de leur diversité de fonction. Nous développons pour cela des approches de génétique fonctionnelle, de biochimie et d’imagerie haute résolution, afin d’identifier et de comprendre la fonction de nouveaux gènes impliqués dans l’assemblage des cils. Nous exploitons principalement le modèle de la drosophile et des modèles cellulaires de souris ou humains. Nos travaux ont des retombées pour la compréhension des maladies associées à des dysfonctionnements de ces organelles.